Ein neues deep-learning-Algorithmus schnell und präzise zu analysieren, verschiedene Arten von genomischen Daten von kolorektalen Tumoren für die genauere Klassifizierung, die helfen könnten, verbessern die Diagnose und damit verbundene therapeutische Optionen, entsprechend der neuen Forschung veröffentlicht in der Zeitschrift Life Science Alliance.
Kolorektale Tumoren sind extrem vielfältig, wie Sie sich entwickeln, erfordern unterschiedliche Medikamente und haben sehr unterschiedliche überlebensraten. Oft, Sie werden klassifiziert in Subtypen auf der Grundlage einer Analyse der gen-expression. „Krankheit ist sehr viel komplexer als nur ein gen,“ sagte Altuna Akalin, Bioinformatik Wissenschaftler, führt in die Bioinformatik-Plattform research-Gruppe am MDC Berlin Institut für Medizinische Systembiologie (BIMSB). „Zu schätzen, die Komplexität, die wir haben, um eine Art des maschinellen Lernens, um wirklich nutzen Sie alle Daten.“
Zu sehen bei zahlreichen Funktionen im genetischen material, einschließlich Genexpression, einzelne Punktmutationen und DNA-Kopie-Zahl, Akalin und Ph. D.-student Jonathan Ronen entwickelt, die Multi-omics Autoencoder Integration Plattform—MAUI kurz.
Wie es funktioniert
Supervised machine learning erfordert normalerweise menschlichen Experten, um die label-Daten und dann mit dem Zug einen Algorithmus, um vorherzusagen, diese Etiketten. Zum Beispiel, um vorherzusagen, Auge Farbe von Bildern von Augen, die Forscher zunächst feed-Algorithmus für Bilder, wo die Augenfarbe ist beschriftet. Der Algorithmus lernt, sich zu identifizieren, verschiedene Auge Farben und können selbstständig analysieren, neue Daten.
Im Gegensatz dazu unüberwachten maschinellen Lernens, nicht um Ausbildung. Ein deep-learning-Algorithmus zugeführt Daten ohne Etiketten und siebt durch die Suche nach gemeinsamen mustern oder repräsentative Funktionen, die als latente Faktoren. Für Beispiel, diese Art von Algorithmus verarbeiten kann Bilder von Gesichtern, die sind nicht beschriftet, die in irgendeiner Weise, dann identifizieren die wichtigsten Merkmale, wie Auge Farben, Augenbrauen Formen, Nase Formen und lächelt.
Als deep-learning-Plattform, MAUI ist in der Lage zu analysieren, die verschiedenen „omics“ – Datensätze und identifizieren die relevanten Muster oder Merkmale, in diesem Fall, gen-sets oder Wege zum Darmkrebs.
Umgliederung von Subtypen?
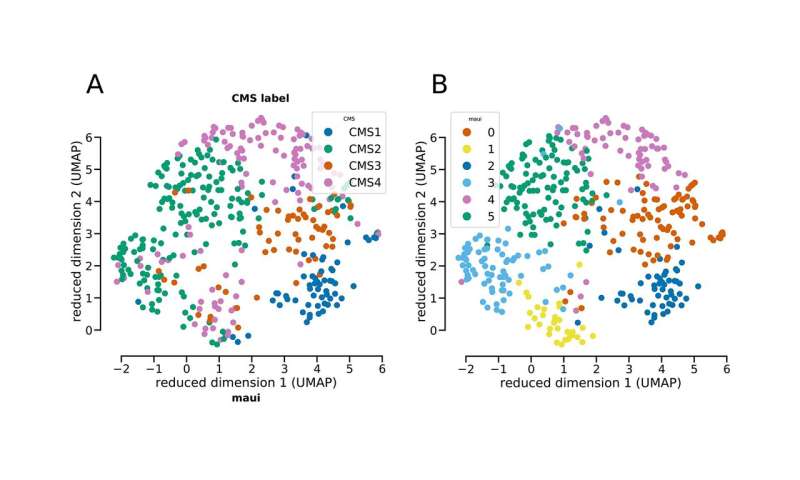
MAUI identifiziert Muster im Zusammenhang mit den vier etablierten Subtypen des kolorektalen Karzinoms, Zuordnung der Tumoren zu den Subtypen mit hoher Genauigkeit. Es ist auch eine interessante Entdeckung gemacht. Die Plattform gefunden, ein Muster, das schlägt einem Subtyp (CMS2) müssen ggf. aufgeteilt werden in zwei separaten Gruppen. Die Tumoren haben unterschiedliche Mechanismen und überlebensraten. Das team schlägt vor, weitere Untersuchungen, um zu überprüfen, ob der Subtyp ist einzigartig oder vielleicht Vertreter der tumor breitet sich aus. Dennoch, es zeigt, dass die macht von der Plattform zu nehmen, alle Daten, anstatt nur die bekannten Gene, die mit einer Krankheit, und produzieren tiefere Einblicke.
„Data science mit komplexen Daten, die schwer zu handhaben sind andere Wege und macht Sinn“, sagte Akalin. „Sie können es füttern alles, was Sie haben, auf die Tumoren, und es findet sinnvolle Muster.“

Schneller, besser
Das Programm war nicht nur präziser, er arbeitet auch viel schneller als andere machine-learning-algorithmen—drei Minuten zu wählen, die aus 100 Muster im Vergleich zu den anderen Programmen, die dauerte 20 Minuten und 11 Stunden.
„Es ist in der Lage zu lernen, um Größenordnungen mehr latenten Faktoren, zu einem Bruchteil der Rechenzeit,“ sagte Jonathan Ronen, der erste Autor der Forschung.
Das team war überrascht, wie schnell das system führt, vor allem, weil Sie nicht über die Verwendung von GPUs zur Beschleunigung von Berechnungen. Dies zeigt, wie extrem gut optimiert der Algorithmus ist, obwohl Sie weiterhin für die Feinabstimmung des Systems.
Die Verbesserung der drug discovery
Das team, das auch die Bayer AG Bioinformatiker Sikander Hayat, angepasst an Ihr Programm zu analysieren Zelllinien genommen von Tumoren gewachsen und in Laboren für die Untersuchung der Effekte von möglichen Drogen-Behandlungen. Jedoch Zelllinien unterscheiden sich auf molekularer Ebene von echten Tumoren in vielerlei Hinsicht. Das team MAUI zu vergleichen Zelllinien, die derzeit für Testzwecke genutzt Dickdarm-Krebs-Medikamente, um zu sehen, wie eng Sie verwandt sind zu echten Tumoren. Fast die Hälfte der Zeilen gefunden wurden, um mehr in Bezug auf andere Zelllinien, als die tatsächlichen Tumoren. Eine Handvoll wurden gefunden, um die besten Linien, die am ehesten repräsentieren die verschiedenen Klassen von CRC-Tumoren.
Beim drug-discovery-Forschung bewegt sich Weg von Zell-Linien, diese Einsicht könnte helfen, maximieren Sie die potentielle Wirkung der Zell-Linie der Forschung, und könnten angepasst werden, für andere Arten von genetischen-basierte drug-testing-tools.
Google und Tumoren
Nun, dass die deep-learning-Plattform für Darmkrebs festgestellt wurde, könnte es verwendet werden, um Daten zu analysieren, für neue Patienten.
„Denken wie eine Suchmaschine“, sagte Akalin.
Ein Arzt könnte die Eingabe des neuen Patienten, die genetischen Daten in MAUI zu finden, die am ähnlichsten ist, um schnell und genau zu klassifizieren und den tumor. Die Plattform könnte beraten, welche Medikamente wurden verwendet, die auf der nächsten übereinstimmenden Tumoren sind und wie gut Sie gearbeitet, wodurch die Vorhersagen Droge Antworten und das überleben von outlook.